Los microorganismos constituyen la mayor porción de biomasa en la Tierra y son ubicuos en el medio ambiente. Coexisten en comunidades microbianas mixtas, cuyas acciones coordinadas contribuyen al sostenimiento de la vida en el planeta. Los inventarios y estudios sobre su diversidad (en términos de abundancia y composición) permiten comprender su distribución en el entorno, su interacción con otros grupos biológicos y su función en la naturaleza. Realizar un inventario sistemático de microorganismos en regiones poco exploradas, como la Amazonia colombiana, no solo genera información valiosa y enriquece los inventarios nacionales de biodiversidad, sino que también proporciona pautas para la conservación y el uso sostenible de estos importantes recursos genéticos.
Conservar los recursos genéticos microbianos de la Amazonia colombiana es fundamental para avanzar en la investigación científica y expandir el conocimiento sobre la biodiversidad, así como para explorar su aplicación en procesos de biorremediación, restauración ecológica y bioprospección.
Equipo técnico
Ana Lucia Noguera, Angie Natalie Díaz, Carolina Díaz, Gladys Cardona, María Camila Escobar y Mayra Hurtado
Palabras clave
Conservación ex situ, recurso genético microbiano, microorganismos endófitos, microorganismos rizosféricos, gen 16S rRNA.
Área geográfica
Guaviare. San José de Guaviare- Estación Experimental El Trueno. Amazonas- Leticia
Resultados
Incremento del conocimiento de la biodiversidad amazónica en todos sus niveles de expresión
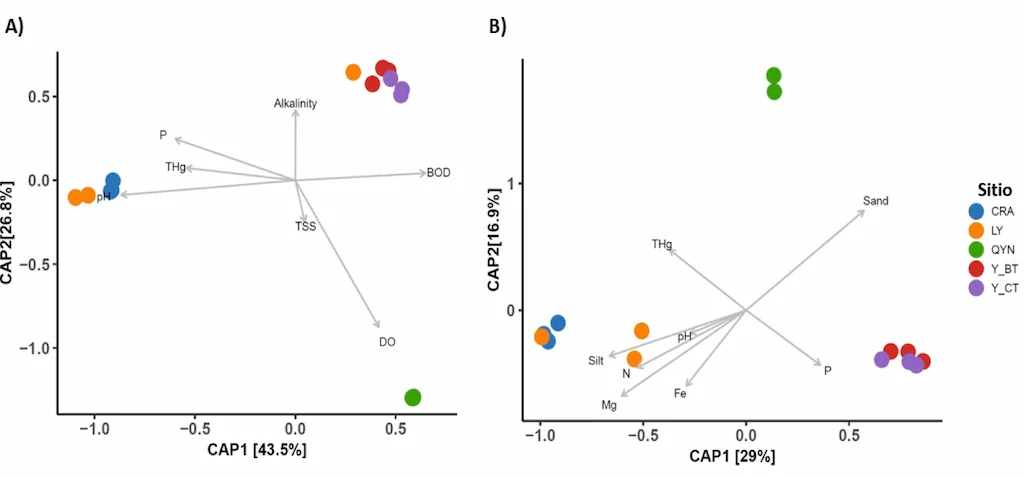
Se estudió la composición de las comunidades bacterianas de muestras de agua y sedimento del sistema hídrico asociado a la quebrada Yahuarcaca en los puntos Colegio Camilo Torres (Y-CT), Bocatoma del acueducto (Y-BT), Nacimiento (balneario Km 8) (QY-N), Lago Yahuarcaca grande (La Playa) (LY) y canal del Rio Amazonas (Isla de la fantasía) (CRA)). El estudio de la diversidad microbiana realizado por el análisis del marcador taxonómico 16S rRNA (región V3-V4) usando la plataforma Quantitative Insights into Microbial Ecology (QIIME2) versión 2019.1., evidencia diferencias en la estructura de las comunidades microbiana entre los sitios de muestreo. En todas las muestras se detectó predominio de Proteobacteria de la familia Burkholderaceae, sin embargo, a nivel de género se detectó que, en la mayoría de las muestras, excepto QYN, predomino el género Polynucleobacter, descritos como organismos de vida libre ubicuas y abundantes en lagos, estanques y arroyos. En muchos sistemas de agua dulce representan más del 10% del bacterioplancton (Jezberová et al., 2010) y están clasificadas ecológicamente como ultramicrobacterias oligotróficas.
En QYN predominó el género Limnohabitans (Proteobacteria, Burkholderaceae), descritos principalmente como organismos con potencial para la fijación de CO2, oxidación del amoniaco y de azufre (Kasalický et al., 2013). Adicionalmente, en todas las muestras se detecta predominio de Actinobacteria, en CRA también se observó alta representación de Cyanobacterias y en QYN de Bacteroidetes. Las diferencias entre la composición de la comunidad bacteriana se asocian a los valores de pH, DBO y oxígeno disuelto (Figura 1A).
La composición bacteriana del sedimento fue dominada por Firmicutes, excepto para el sitio QYN, en el cual Actinobacteria fue el filo más abundante, Proteobacteria, Chloroflexi, y Acidobacteria fueron los otros filos con mayor representación en las comunidades de sedimento, variando su dominancia de una muestra a otra. Se resalta que en una de las muestras de LY se encontró la mayor cantidad de Nitrospirae. Al observar el comportamiento de las comunidades bacterianas de sedimento, se encontró que el pH, Mg, nitrógeno, hierro y el contenido de limo estarían más relacionados con la composición de las comunidades bacterianas de CRA y LY, también estás fueron las que mayor relación positiva presentaron con el contenido de mercurio. Asimismo, se encontró que el fósforo estuvo más relacionado con las comunidades bacterianas de Y_BT y Y_CT (Figura 1B).
Figura 1. Análisis de ordenación canónica (CAP). A) Agua B) Sedimento. Las variables utilizadas resultaron de la selección según correlación entre ellas.
Diversidad de microorganismos endófitos y rizosféricos de especies vegetales de plantas de interés ecológico y agroforestal de la región amazónica
Se analizó la diversidad de microorganismos endófitos y rizosféricos de las especies vegetales Euterpe precatoria (Asaí), Minquartia guianensis (Cuyubí) y Cedrelinga catenaeformis (Achapo), plantas de interés ecológico y agroforestal de la región amazónica. La comunidad de bacterias endófitas de la raíz de Asía en su mayoría (61,1%) no fue identificada taxonómicamente indicando la enorme diversidad sin explorar. Entre los grupos detectados están Nocardia (14,5%), Rhizobium (13,6%) y Dickeya (8,4%). En Cuyubi se encontró predominio del género Rhizobium (59,7%), seguido de un 28,9% de secuencias (ASV) sin asignación taxonómica, Dickeya (6,3 %) y Nocardia (3,3 %). En la raíz de Achapo se reporta predominio de organismos de la clase Alfaproteobacteria del género Rhizobium (80,45%), seguido de 13,5% de ASV sin clasificación taxonómica, Nocardia (2,8%) y Dickeya (2,03%).
El estudio metagenómico de organismos rizosféricos que implica conocer tanto asignación taxonómica como genes asociados a rutas metabólicas, evidencia que a nivel taxonómico casi el 60% de las secuencias obtenidas no logran ser clasificadas, lo cual confirma la enorme diversidad microbiana aún desconocida asociada a plantas Amazónicas. Las secuencias clasificadas indican predominio del Dominio Bacteria (91%) asociados al filo Proteobacteria de la clase Alfaproteobacteria del orden Rhizobiales y Betaproteobacteria del género Burkholderia, Actinobacteria y Acidobacteria. En menor cantidad se encontraron secuencias asociadas al Dominio Archaea (0,4- 0,9 %) y a Virus (8%). A pesar de realizar la anotación con bases de datos que incluyen el Dominio Eukarya, no se encontraron secuencias asociadas lo cual podría ocurrir por problemas durante la lisis en los procesos de extracción de ADN.
Los grupos predominantes están relacionados con procesos de promoción de crecimiento vegetal, fijación de CO2 y reciclaje de carbono. Estos datos se corroboran con los datos funcionales deducidos de la anotación de genes entre los cuales se detectan rutas metabólicas asociadas a la degradación de carbono, principalmente de compuestos poliméricos como la celulosa y el glucano. Procesos de fijación anaerobia y aerobia de CO2. Metabolismo del nitrógeno que incluye oxidación de amoniaco a nitritos y oxidación de nitritos a nitrato, así como, procesos de reducción disimilatoria de nitrito. Se encontraron genes asociados a metagenogénesis.
Los datos obtenidos generan información sobre el contexto ecológico asociado a los microorganismos rizosféricos de plantas Amazónicas de interés en procesos agroforestales y de restauración. Estos datos podrían ser la base para la formulación de propuestas orientadas a mejorar la resistencia de las plantas a factores abióticos como el estrés hidrico y a la promoción de crecimiento vegetal de plantulas empleadas en los procesos de restauración ecológica.
Fichas del capítulo I